- Avant propos, pour aller plus loin
- Introduction
- C’est parti
- Reproductibilité
- Pour aller plus loin
- Bêtisier
Avant propos, pour aller plus loin
Pour voir des exemples plus avancés vous pouvez jeter un coup d’oeil à cette tentative de reproduction d’un article analysant des données à propos d’un tournoi de starcraft.
Introduction
L’objectif de cet article est de démonter qu’il est possible de traiter des données scientifiques en ligne de commande, via des outils traditionnellement disponibles sur les distributions linux et/ou des outils relativement simples se manipulant bien dans un terminal. On ne fera rien de très complexe parce que
- Je ne sais pas le faire, je ne suis pas data-scientist
- Les outils que nous verrons sont volontairement assez simples
Nous allons traiter des fichiers TSV1 parce que j’ai constaté que de très nombreux projets scientifiques font du traitement de données tabulaires. Si l’on parvient à faire des choses intéressantes avec on répond donc à de nombreux besoins. Il est fréquent que les chercheureuses dégainent des outils très complexes, capables de faire bien plus que ce que l’on va voir, pour faire de opérations très simples. L’idée est ici de rendre plus tangible ce qu’il est possible de faire sans déployer ces outils pour éviter de vider son bol de céréales avec une cuillère de deux mètres - 524Ko.
Je vais introduire beaucoup de commandes sans les expliquer en profondeur.
Nombreuses d’entre elles ne seront utilisées que d’une seule façon ou dans un
seul contexte alors qu’elles regorgent d’options. Je vous incite fortement à
être curieux et curieuses et à lire les manuels des ces commandes en tapant
man commande, commande --help ou encore info commande pour en apprendre
plus. Quand j’écris “cette commande sert à/fait cela”, il est très rare qu’elle
ne serve qu’à cela.
Presque tout ce que je vais faire peut ête reproduit d’autres manières, certaines possiblement meilleures. C’est le propre des interfaces en ligne de commande UNIX. Si vous voulez proposer des alternatives n’hésitez pas à contacter le collectif par mail ou en présentiel, je modifierai l’article avec plaisir.
Finalement, beaucoup de tabulations seront utilisés dans le code présenté dans
cet article. Malheureusement elles ressemblent à des espaces dans les blocs de
code, je vais réfléchir à un moyen d’y remédier. En attendant vous pouvez
lire cet article en markdown dans vim en faisant curl
http://katzele.netlib.re/articles/datascience-cli/index.md | vim - et faire
apparaître les tabulations avec la commande vim set listchars=tab:\ \ \|.
Pour comprendre ce que cela fait vous pouvez consulter l’aide :help listchars.
C’est parti
CSV vs TSV
Prenons ce fichier (de 500Ko) pour exemple de jeu de donnée : https://raw.githubusercontent.com/datasets/population/main/data/population.csv
Je vais d’abord le convertir en TSV. Les raisons pour lesquelles je préfère le TSV sont résumées dans cet article d’une équipe d’eBay.
Un moyen de convertir notre fichier CSV en TSV serait de simplement remplacer
toutes les occurrences de , par des tabulations. L’outil tr2 permet de
traduire un ensemble de caractères en un autre. Le début de population.csv
contient :
Country Name,Country Code,Year,Value
Aruba,ABW,1960,54608
Aruba,ABW,1961,55811
Aruba,ABW,1962,56682
Aruba,ABW,1963,57475
Aruba,ABW,1964,58178
Aruba,ABW,1965,58782
Aruba,ABW,1966,59291
Aruba,ABW,1967,59522
Aruba,ABW,1968,59471
Si l’on fait `tr ‘,’ ‘\t’ < population.csv > population.tsv’3 :
Country Name Country Code Year Value
Aruba ABW 1960 54608
Aruba ABW 1961 55811
Aruba ABW 1962 56682
Aruba ABW 1963 57475
Aruba ABW 1964 58178
Aruba ABW 1965 58782
Aruba ABW 1966 59291
Aruba ABW 1967 59522
Aruba ABW 1968 59471
on obtient bien un fichier TSV. Ici la tâche a été très simple puisque nos données ne contiennent jamais de virgules ni aucun échappement en particulier. Résoudre cette tâche à coup sûr est moins triviale qu’il n’y paraît. J’opte donc généralement d’utiliser csv2tsv des outils tsv-utils.
csv2tsv population.csv > population.tsv
Compter des entrées
Première question, combien y a-t-il d’entrée dans notre fichier / base de
donnée ? wc est un outil permettant de compter les lignes d’un fichier :
wc -l population.tsv
16401 population.tsv
Si on ne passe pas le fichier en argument mais directement dans stdin wc n’affiche
pas le nom du fichier en sortie
wc -l < population.tsv
16401
Si vous avez un jeu de donnée contenant pleins de TSV et que vous voulez exécuter wc sur plusieurs de ces fichiers :
wc -l population.tsv population.tsv
16401 population.tsv
16401 population.tsv
32802 total
wc vous fera gentiment un affichage avec le total et le sous total par fichier.
Admettons que nous voulions maintenant regarder à l’intérieur du fichier. Il est souvent intéressant de n’afficher que
le début ou la fin pour éviter d’être submergé·e par un mur de texte. head et tail servent à cela :
head population.tsv
Country Name Country Code Year Value
Aruba ABW 1960 54608
Aruba ABW 1961 55811
Aruba ABW 1962 56682
Aruba ABW 1963 57475
Aruba ABW 1964 58178
Aruba ABW 1965 58782
Aruba ABW 1966 59291
Aruba ABW 1967 59522
Aruba ABW 1968 59471
Si jamais vous voulez un affichage où les colonnes sont alignées vous pouvez
utiliser column en lui disant que le séparateur est la tabulation4 :
head population.tsv | column -ts ' '
Country Name Country Code Year Value
Aruba ABW 1960 54608
Aruba ABW 1961 55811
Aruba ABW 1962 56682
Aruba ABW 1963 57475
Aruba ABW 1964 58178
Aruba ABW 1965 58782
Aruba ABW 1966 59291
Aruba ABW 1967 59522
Aruba ABW 1968 59471
Sinon tsv-utils fournit son propre outil tsv-pretty qui semble faire la même chose mais probablement plus/mieux (j’ai pas vérifié) :
head population.tsv | tsv-pretty
Country Name Country Code Year Value
Aruba ABW 1960 54608
Aruba ABW 1961 55811
Aruba ABW 1962 56682
Aruba ABW 1963 57475
Aruba ABW 1964 58178
Aruba ABW 1965 58782
Aruba ABW 1966 59291
Aruba ABW 1967 59522
Aruba ABW 1968 59471
Lister des valeurs
Disons que l’on souhaite obtenir la liste de tous les pays présents dans ce jeu
de données. D’abord restreignons nous à la colonne qui nous intéresse, la
première, avec cut :
head population.tsv | cut -f1
Country Name
Aruba
Aruba
Aruba
Aruba
Aruba
Aruba
Aruba
Aruba
Aruba
Pas besoin de dire à cut quel est le délimiteur, la tabulation est celui par
défaut. On lui dit à l’aide de l’argument -f quelle colonne on veut (la
première). Notre jeux de donnée affiche l’évolution des populations des pays
par années, on se retrouve donc avec pleins de duplicas. Pour résoudre ce souci
on peut utiliser uniq. En le faisant sur la totalité du fichier cette fois -ci :
cut -f1 population.tsv | uniq
Country Name
Aruba
Africa Eastern and Southern
Afghanistan
Africa Western and Central
Angola
Albania
Andorra
Arab World
United Arab Emirates
Argentina
...
On peut aussi savoir rapidement combien de pays sont concernés en combinant avec wc :
cut -f1 population.tsv | uniq | wc -l
266
Ce qui semble vraiment beaucoup. Effectivement notre jeux de donnée contient
des regroupements de pays tels que “Europe & Central Asia”. Cette capacité à combiner
des commandes est l’une des propriétés vraiment puissantes de l’utilisation du pipe (|)
dans le traitement de données. D’ailleurs c’est tellement puissant que R l’a reproduit
tel quel dans son langage. On remarque que le pays “United Arab Emirates” apparaît avant
le pays “Argentina”. C’est l’occasion de voir comment trier les données. On utilise la
commande sort :
cut -f1 population.tsv | uniq | sort
Afghanistan
Africa Eastern and Southern
Africa Western and Central
Albania
Algeria
American Samoa
Andorra
Angola
Antigua and Barbuda
Arab World
Argentina
...
On voit que l’entrée des Emirats Arabes Unis apparaît désormais plus loin.
Filtrer le fichier
Revenons à nos données. Admettons que l’on ne veuille retenir que l’années
2002. On va filtrer le fichier avec grep :
grep "2002" population.tsv | tsv-pretty
Aruba ABW 2002 91781
Africa Eastern and Southern AFE 2002 422741118
Afghanistan AFG 2002 21000256
Africa Western and Central AFW 1996 242200260
Africa Western and Central AFW 2002 284952322
Angola AGO 2002 17516139
oups petit souci, on constate que pour le regroupement “Africa Western and
Central” deux années on été retenues. Pourquoi ? Parce qu’en 1996 la population
de cette région s’élevait à 2422002260 personnes. grep ne sait pas ce
qu’est une colonne. Il y a plusieurs solutions à cela :
- En utilisant
grep, on fait non plus une recherche sur2002mais2002(tab2002tab)
Puisqu’il n’y a pas de tabulation dans les données :
grep " 2002 " population.tsv | tsv-pretty
Aruba ABW 2002 91781
Africa Eastern and Southern AFE 2002 422741118
Afghanistan AFG 2002 21000256
Africa Western and Central AFW 2002 284952322
Angola AGO 2002 17516139
Albania ALB 2002 3051010
- On utilise un autre outil qui “comprend” les colonnes.
Par exemple awk (je ne rentre pas dans les détails pour le moment) : awk -F'
' '$3=="2002"' population.tsv. $3 désigne la troisième colonne, on vérifie
son égalité avec “2002”. Par défaut si le test est vrai la ligne est affichée
dans awk. Un autre exemple serait, toujours dans la suite tsv-utils, l’outil
tsv-filter, tsv-filter -H --eq 3:2002 population.tsv.
Maintenant que l’on a les données pour l’année 2002, comment savoir, par exemple
quel est le pays / région la plus peuplée ? On ressort sort de notre besace.
grep " 2002 " population.tsv | sort -t' ' -nk4
Tuvalu TUV 2002 9609
Nauru NRU 2002 10351
Palau PLW 2002 19851
Turks and Caicos Islands TCA 2002 20598
British Virgin Islands VGB 2002 21288
Gibraltar GIB 2002 27892
San Marino SMR 2002 27969
Sint Maarten (Dutch part) SXM 2002 30777
Avec -t on dit à sort quel est le délimiteur (par défaut c’est n’importe
quelle transition d’un caractère non “blanc” vers un caractère “blanc”) puis
avec -n que l’on veut trier des chiffres (par défaut du texte) puis avec
-k4 que l’on veut trier sur le 4ème champ (la population). Avec un -r
on trie par ordre décroisant (par défaut par ordre croissant comme on le
constate au dessus).
grep " 2002 " population.tsv | sort -t' ' -nrk4
World WLD 2002 6308092739
IDA & IBRD total IBT 2002 5243235123
Low & middle income LMY 2002 5167898454
Middle income MIC 2002 4744040472
...
Afficher une courbe
Nous souhaitons maintenant regarder l’évolution de la population d’un pays
donné et tenter de la visualiser. Prenons la France pour exemple. Nous allons
trier avec grep puis utiliser gnuplot pour le graph. J’aime bien
gnuplot parce que malgré ses commandes et sa logique parfois un peu
confuse5, c’est un logiciel qui s’utilise bien en ligne de commande.
grep "France" population.tsv | cut -f3,4 | gnuplot -p -e "plot '-'"
On a utilisé la cut pour retenir cette fois plusieurs colonnes, celle des
années et celle de la population en listant leurs numéros séparés par des
virgules. Le résultat donne ceci :
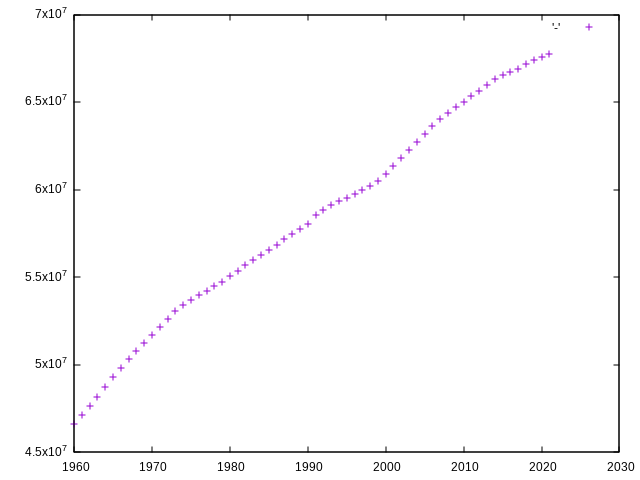
J’espère que vous appréciez la puissance de la combinaison d’outils simples en ligne de commande pour des traitements modestes comme celui-ci. En quelques caractères et en installant uniquement gnuplot6 on peut filtrer un gros fichier texte et grapher des valeurs numériques en cinq centièmes de secondes (sur ma machine).
Les paramètres par défaut de gnuplot pour un script aussi simple (plot '-')
donnent des résultats insatisfaisant si l’on souhaitait publier quelque chose de
sérieux puisqu’il n’y a pas de titre, la légende ne fait pas sens, les axes ne
sont pas labellisés, la population est exprimée en notation scientifique.
Gnuplot permet de modifier tout cela mais ce n’est pas l’objet de cet article.
Je vous renvoie vers la documentation. Bonne chance. Pour l’anecdote les sorties
de gnuplot sont nombreuses (png, svg, pdf, jpg, html etc) mais l’une d’entre
elle est plus inattendue que les autres, c’est la sortie ascii. Il faut exécuter
la commande set term dumb, on obtient alors :
grep "France" population.tsv | cut -f3,4 | gnuplot -p -e "set term dumb;plot '-'"
7e+07 +----------------------------------------------------------------+
| + + + + + + |
| AAAAA A |
| AAAAA |
6.5e+07 |-+ AAA +-|
| AAA |
| AA |
| AAA |
6e+07 |-+ AAAA +-|
| AAAA |
| AAAA |
| AAAAA |
5.5e+07 |-+ AAAA +-|
| AAAA |
| AAA |
| AAA |
5e+07 |-+ AA +-|
| AAA |
|AA |
| + + + + + + |
4.5e+07 +----------------------------------------------------------------+
1960 1970 1980 1990 2000 2010 2020 2030
Voilà, marrant.
Faire un filtre un peu plus avancé
Nous voulons dorénavant savoir combien il existe de pays (et de régions puisque notre jeu de donée ne fait pas la différence) de plus de 50M d’habitant·e·s en 2020 :
grep " 2020 " population.tsv | cut -f4 | grep -E "^([5-9][0-9]{7}|[1-9][0-9]{8,})$" | wc -l
73
On filtre sur 2020, on ne conserve que la colonne de la population et on filtre sur les nombre supérieurs ou égaux à 50M pour finalement compter le nombre de ligne en résultat. J’introduis ici quelque chose d’assez complexe mais incroyablement puissant, les expressions régulières. Je ne vais pas les expliquer en détail mais celle que nous avons utilisé se comprend comme ça :
[5-9][0-9]{7}= 5,6,7,8 ou 9 suivi de n’importe quels sept chiffres (autrement dit tous les nombres entre 50M et 99 999 999)[1-9][0-9]{8,}= 1,2,3,4,5,6,7,8 ou 9 suivi de n’importe quels huit chiffres (autrement dit 100M et au delà)
Mettre les deux entre parenthèses séparées par un pipe | veut dire l’un ou
l’autre. Le ^ veut dire “début de la ligne”, le $ veut dire la fin, c’est
pour garantir de parser le nombre en entier. Je vous recommande très fortement
d’apprendre à utiliser les regex. Un bon bouquin serait Mastering Regular
Expressions que je peux vous prêter en français (dans une vieille édition) ou
que vous pouvez trouver sur internet en cherchant bien. Si on estime cela
utile/nécessaire on peut aussi utiliser tsv-filter --ge 4:50000000 qui
est un peu moins ésotérique. Si l’on voulait conserver la colonne des pays,
pour grapher leurs populations par exemple, il faudrait modifier la regex
pour lui dire de ne s’intéresser qu’à la dernière colonne ce qui permet
de sauter le cut qui retire l’info que l’on veut :
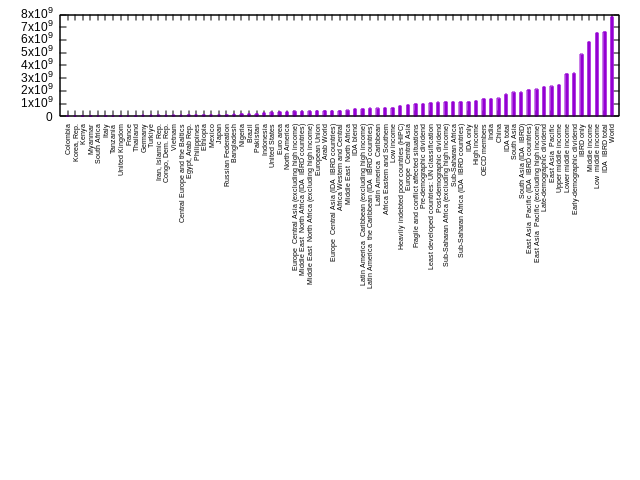
grep " 2020 " population.tsv |
grep -E "([5-9][0-9]{7}|[1-9][0-9]{8,})$" |
cut -f1,4 |
sort -t' ' -nk2 > data
gnuplot -p -e "set style fill solid;
set xtics rotate by 90 right;
set datafile separator ' ';
set xtics font ',5';
plot 'data' using 2:xticlabels(1) with histogram notitle"
Pour une raison qui m’est encore inconnue gnuplot ne veut pas générer la totalité de cet histogramme. Je vais chercher pourquoi, en attendant je vous met le résultat bugué.
Je crois avoir trouvé. Il se trouve que lorsque gnuplot lit dans stdin il s’arrête lorsqu’il voit un
esur une nouvelle ligne. Ici le pays qui suivait l’europe centrale était l’Egypte, commençant donc par une. Je trouve cela surprenant que le fonctionnement par défaut ne soit pas de s’arrêter à uneseul comme pour un here-doc mais soit. Pour y remédier je dépose donc les données dans un fichier. Le code au dessus a été corrigé.
Des sommes, des moyennes, des écarts type etc
Calculons maintenant des valeurs statistiques. Par exemple, quel serait la somme de la population de la France, l’Allemagne et l’Italie en 1973 ?
grep -E "(France|Germany|Italy).+ 1973 " population.tsv
Germany DEU 1973 78936666
France FRA 1973 53053660
Italy ITA 1973 54751406
grep -E "(France|Germany|Italy).+ 1973 " population.tsv |
cut -f4 |
paste -s -d'+'
78936666+53053660+54751406
grep -E "(France|Germany|Italy).+ 1973 " population.tsv |
cut -f4 |
paste -s -d'+' |
bc -l
186741732
On utilise ici paste pour coller les lignes de nos données une à une avec le
délimiteur + ce qui créé une expression que bc (une calculette en ligne de
commande) peut comprendre. Si l’on voulait faire ce traitement pour toutes les
années :
cut -f3 population.tsv | tail -n+2 | sort -u |
xargs -d'\n' -n1 sh -c 'printf "$1 ";
grep -E "(France|Germany|Italy).+ $1" population.tsv |
cut -f4 |
paste -s -d"+" |
bc -l
' -- |
gnuplot -p -e "plot '-'"
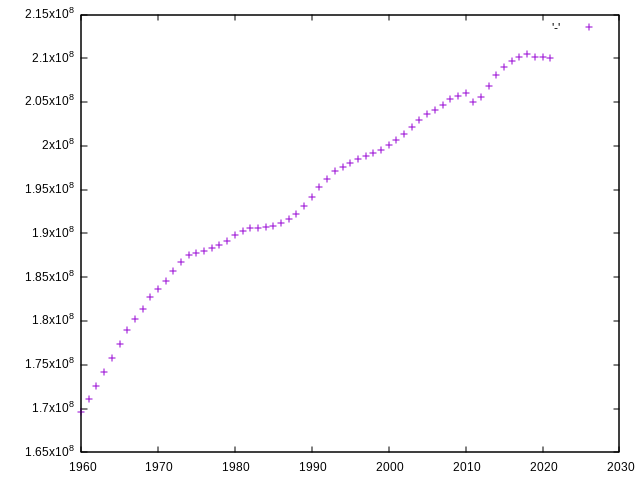
Je reconnais que ça commence à être un peu velu. Sur la première ligne on
récupère la liste des années. Le tail permet d’enlever la première ligne
(l’entête) et le sort -u de retirer les doublons. Ensuite on utilise xargs
pour appeler la commande qui nous donnait la somme des populations pour une
année donnée successivement sur toutes les années que la première ligne nous
donne. Il faut bien sûr remplacer le 1973 par un $1 qui est une variable
qui sera remplacée par l’année. Il y a beaucoup plus à expliquer à propos
d’xargs mais je vais en rester là.
Il est à noter que cet exemple est relativement peu efficace. La commande termine en deux dixièmes de secondes sur ma machine, ce qui me semble tout à fait respectable, mais sur un gros fichier ce temps d’exécution aurait explosé. De plus l’écriture de la commande n’est pas aisée. Si vous rencontrez des limites rédactionnelles ou de performance dans ce genre de cas c’est peut-être que vous devriez utiliser d’autres outils. Par exemple, en utilisant la suite tsv-utils :
tsv-filter --regex 1:"France|Germany|Italy" population.tsv |
tsv-summarize --sum 4 --group-by 3 |
gnuplot -p -e "plot '-'"
tsv-filter ne garde que les lignes dont la première colonne (1:"France…)
match la regex (–regex 1:…) qui match soit la France soit l’Allemagne soit
l’Italie (1:"France|Germany|Italy”. Ensuite tsv-summarize fait une somme de
la quatrième colonne (--sum 4) en groupant par années (--group-by 3).
Il faut reconnaître que c’est un peu plus facile à écrire et à lire. C’est
aussi dramatiquement plus efficace puisque la commande termine en 14 centièmes
de secondes soit presque 20 fois moins que celle avec xargs. Cela pour deux
raisons principales. Premièrement les tsv-utils ont été bien
optimisés pour faire de la lecture de fichiers tsv et tsv uniquement
là où les autres outils sont souvent plus généralistes et ne peuvent donc
pas se permettre certaines optimisations. Deuxièmement l’algorithme interne
de tsv-summarize permettant de faire une opération en groupant sur une
autre variable est terriblement plus efficace que de bêtement refiltrer
sur chacune des années avec un grep etc. Cette dernière technique
ouvre un nouveau processus à chaque année ce qui constitue un overhead
significatif. Si vous avec besoin de plus de performance et où s’il est
vraiment important d’exprimer ce que vous voulez plus simplement alors
tsv-utils pourrait vous être utile7.
tsv-summarize sait aussi calculer des statistiques assez classiques telles que
la moyenne, la médiane, l’écart type etc. Les tsv-utils savent aussi adresser
les colonnes par leurs noms si un entête existe. A chaque fois que l’on a
utilisé des chiffres on aurait pu utiliser le nom de la colonne.
Et des jointures ?
Et si l’on veut faire des jointures ? Admettons que notre fichier soit séparé en deux, d’un côté nous avons les codes des pays, les années et les valeurs, de l’autre nous avons la correspondance entre les codes des pays et leur noms complets.
head correspondance.tsv sans-pays.tsv
==> correspondance.tsv <==
Country Name Country Code
Aruba ABW
Africa Eastern and Southern AFE
Afghanistan AFG
Africa Western and Central AFW
Angola AGO
Albania ALB
Andorra AND
Arab World ARB
United Arab Emirates ARE
==> sans-pays.tsv <==
Country Code Year Value
ABW 1960 54608
ABW 1961 55811
ABW 1962 56682
ABW 1963 57475
ABW 1964 58178
ABW 1965 58782
ABW 1966 59291
ABW 1967 59522
ABW 1968 59471
...
Alors on peut faire une jointure avec
join --header -t' ' -1 2 -2 1 correspondance.tsv sans-pays.tsv
Country Code Country Name Year Value
ABW Aruba 1960 54608
ABW Aruba 1961 55811
...
On saute les premières lignes puisque ce sont des header (--header), on
choisit la tabulation comme délimiteur (-t' '), dans le premier fichier on
joint sur le second champ (le code, -1 2), dans le second sur le premier (-2
1) et on passe les fichier en argument. tsv-utils a un équivalent un peu plus
versatile qui pour le même besoin s’utiliserait de la sorte :
tsv-join -H --filter-file correspondance.tsv -k 1 -a 2 sans-pays.tsv
Country Code Year Value Country Name
ABW 1960 54608 Aruba
ABW 1961 55811 Aruba
ABW 1962 56682 Aruba
ABW 1963 57475 Aruba
...
Attention, je crois qu’il faut que la colonne sur laquelle faire la jointure soit la même dans les deux fichiers. Limitation un peu étrange mais soit.
Compter les occurrences des valeurs d’une colonne
Pour continuer les exmples prenons le jeu de donnée ici : https://github.com/datablist/sample-csv-files/raw/main/files/people/people-2000000.zip. Attention le fichier pèse 66Mo zippé, 255M dézippé. Je prends un gros fichier comme celui-ci pour vérifier que ce que l’on fait est au moins un peu performant. Vous pouvez télécharger une version plus petite sur la page du dépôt github.
Convertissons le en TSV et regardons ce qu’il y a dedans :
csv2tsv people.csv > people.tsv;head people.tsv | tsv-pretty
Index User Id First Name Last Name Sex Email Phone Date of birth Job Title
1 4defE49671cF860 Sydney Shannon Male tvang@example.net 574-440-1423x9799 2020-07-09 Technical brewer
2 F89B87bCf8f210b Regina Lin Male helen14@example.net 001-273-664-2268x90121 1909-06-20 Teacher, adult education
3 Cad6052BDd5DEaf Pamela Blake Female brent05@example.org 927-880-5785x85266 1964-08-19 Armed forces operational officer
4 e83E46f80f629CD Dave Hoffman Female munozcraig@example.org 001-147-429-8340x608 2009-02-19 Ship broker
5 60AAc4DcaBcE3b6 Ian Campos Female brownevelyn@example.net 166-126-4390 1997-10-02 Media planner
6 7ACb92d81A42fdf Valerie Patel Male muellerjoel@example.net 001-379-612-1298x853 2021-04-07 Engineer, materials
7 A00bacC18101d37 Dan Castillo Female billmoody@example.net (448)494-0852x63243 1975-04-09 Historic buildings inspector/conservation officer
8 B012698Cf31cfec Clinton Cochran Male glenn94@example.org 4425100065 1966-07-19 Engineer, mining
9 a5bd11BD7dA1a4B Gabriella Richard Female blane@example.org 352.362.4148x8344 2021-09-02 Wellsite geologist
Bon c’est pas facile à lire parce que les lignes sont vraiment longues mais soit. Si vous n’utilisez pas tsv-utils mais que vous voulez voir rapidement quels sont les chiffres qui correspondent aux entêtes vous pouvez faire :
head -n1 people.tsv | tr ' ' '\n' | nl
1 Index
2 User Id
3 First Name
4 Last Name
5 Sex
6 Email
7 Phone
8 Date of birth
9 Job Title
Imaginons que nous voulions compter le nombre de personnes identifiées comme “Male” dans ce fichier :
cut -f5 people.tsv | sort | uniq -c
1000505 Female
999495 Male
1 Sex
On ne retient que la colonne pertinente avec cut, on la tri pour que tous les
occurrences identiques soient les unes à côté des autres puis on les compte
avec uniq en lui précisant d’afficher le nombre d’occurrence en utilisant
-c. Il est nécessaire de trier les données avant de pouvoir les compter avec
uniq. uniq est très bête, il ne sait pas tenir les comptes d’une valeur à
une autre.
On voit qu’il y a une occurrence de “Sex”, c’est l’entête que se ballade. Si vous ne
la voulez pas on peut initialement la supprimer avec tail -n+2.
Cette commande s’exécute en 1,4s. Certes le fichier est très gros mais c’est
aussi parce que cette nécessité de trier la colonne avant de compter est
coûteuse. Deux solutions à ça. Premièrement, donner un plus gros buffer à
sort avec --buffer-size=NM où N est un entier. Cela aide mais n’est pas
magique non plus. Si les performances sont vraiment cruciales alors je
suggère de dégainer à nouveau tsv-utils.
tsv-summarize --count --group-by 5 people.tsv
Sex 1
Male 999495
Female 1000505
Cette commande s’exécute en 0,3s. Autre avantage de tsv-summarize dans ce cas
est qu’il renvoie le résultat sous forme d’un TSV qu’on peut donc continuer à
piper facilement. Le résultat d’uniq doit être remanié. J’en donne un exemple
juste après. Pour un exemple similaire mais avec un peu plus de préparation,
imaginons que nous souhaitons grapher la fréquence des années de naissance des
personnes :
tail -n+2 people.tsv | cut -f8 | cut -d'-' -f1 |
sort -n | uniq -c |
sed -E 's/ +([0-9]+) ([0-9]+)/\2 \1/' |
gnuplot -p -e "plot '-'"
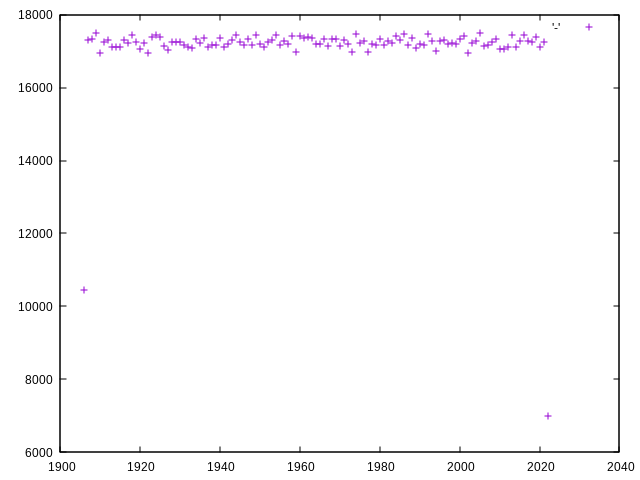
Il semblerait que ce jeu de données ait été généré aléatoirement pour que la répartition soit aussi régulières. Avec tsv-summarize :
cut -f8 people.tsv | cut -d'-' -f1 | tail -n+2 |
tsv-summarize --count -g 1 |
gnuplot -p -e "plot '-'"
Cela évite l’appel à sed pour remanier la sortie d’uniq et va plus de deux
fois plus vite.
Reproductibilité
J’espère avoir fait la démonstration, même courte, qu’il est très facile, rapide et performant d’effectuer des traitements simples sur des gros volumes de données à l’aide des commandes de bases du monde Unix. En plus de cette propriété je pense qu’évoluer aussi longtemps que possible dans ce monde logiciel là est bénéfique pour la reproductibilité de résultats scientifiques.
Les commandes majoritairement utilisées ici sont toutes :
- Très anciennes
Être un vieux logiciel n’est pas un avantage pour la repro en soit mais cela témoigne d’une certaine longévité jusque là qui permet d’être relativement optimiste sur la possibilité qu’il soit là à l’avenir. Les commandes que l’on a vu ont pour la plupart 20/30/40 ans et survivent avec relativement peu de maintenance. De plus, étant déjà fondamentales à l’époque et le temps ayant fait son affaires, elle se sont immiscées partout ce qui diminue les chances qu’elles changent ou pire disparaissent.
- Très stables
Du fait que chaque commande ne sache faire qu’un ensemble relativement restreint de choses et soient isolées les une des autres, elles sont assez rarement mises à jour. Ainsi les chances pour qu’une mise à jour vienne modifier le comportement de votre code ou casser votre environnement sont presque nulles. Dans le milieu on dit que ce sont des logiciels “matures”.
- Très peu buguées
Des décennies de développement et d’usages ont fait de ces commandes des logiciels contenant peu de bugs. La présence de bugs n’est évidemment pas souhaitable dans un contexte où l’on cherche à obtenir des résultats fiables et à les reproduire.
- Généralement disponibles par défaut
Étant historiquement8 le fondement de “l’espace utilisateurice” des systèmes Unix, ces commandes sont naturellement (presque ?) toujours disponibles sur une installation fraîche des systèmes héritier d’Unix. Rien besoin d’installer, pas besoin d’une connexion internet, pas besoin de configurer quoi que soit. En gros, dépendre de ces commandes revient plus ou moins à dépendre de la chaîne de développement et de de distribution qui vous permet d’avoir un système d’exploitation sur votre machine. Or vous ne feriez rien de ce genre sans OS de toute façon donc vous consentez à pratiquement aucune dépendance supplémentaire en utilisant ces logiciels. Je caricature mais la documentation pour permettre de reproduire l’environnement de développement nécessaire à l’obtention des mêmes résultats se limiterait alors presque à “Un OS Linux en état de fonctionnement”. Entre ça et lister les versions précises des dizaines de paquets Python ou R installés mon choix est vite fait.
- Relativement simples dans leurs implémentations
Ces commandes sont des logiciels assez simples relativement à d’autres
outils pour manipuler des données. Cela rend leur maintenance plus simple ce
qui favorise leur pérennité. De plus, puisqu’elles sont toutes indépendantes
les une des autres, la disparition d’une n’implique pas forcément la
disparition d’une autre. Je précise “relativement” puisqu’en réalité, avec
le temps et pour des raisons plus ou moins légitimes, les variantes les plus
répandues de ces commandes sont devenues assez complexes. Par exemple sort
fait :
curl -Ls https://github.com/coreutils/coreutils/raw/master/src/sort.c | wc -l
4847
4847 lignes de C9. Trier n’est pas un problème trivial mais il est possible
de faire moins. Par exemple le projet busybox a réimplémenté sort en :
curl -Ls https://git.busybox.net/busybox/plain/coreutils/sort.c | wc -l
684
684 lignes. Cela dit, bien que je puisse imaginer que le projet GNU ait pris un peu trop de poids “inutile” avec le temps il est manifeste qu’une partie de tout ce code participe aux bonnes performances. Par exemple :
time -f '%e' cut -f5 people.tsv | sort | uniq -c
1.22
1000505 Female
999495 Male
1 Sex
time -f '%e' busybox cut -f5 people.tsv | busybox sort | busybox uniq -c
1.73
1000505 Female
999495 Male
1 Sex
On voit que pour ce traitement les versions busybox sont environ 30% plus lentes.
- Relativement performantes et accessibles via des interfaces simples et performantes
Dans l’ensemble ces commandes sont performantes prises individuellement. Cela permet de diminuer la charge sur le matériel et ainsi permettre de faire tourner les traitements sur un plus grand nombre de machine. Cela permet à plus de personnes y compris sur des machines peu puissantes et peu coûteuse de produire ou reproduire le traitement. Ces outils ont également l’élégance d’être accessible via des interfaces très simples, en ligne de commande, qui peuvent tourner sur des machines peu puissantes. De nombreux outils de traitement de données beaucoup plus complexes sont également accessibles en ligne de commande et je pense qu’ils devraient, au moins initialement, être enseignés via cette interface. Pour pouvoir tourner sur plus de machines mais aussi pour inciter à l’utilisation des commandes que l’on a vu dans cet article. On ne dégaine alors l’outil plus complexe que lorsqu’il le faut, au détour d’un pipe, exactement comme nous l’avons fait avec les tsv-utils ou gnuplot.
Évidemment ce n’est pas magique et pour des besoins un peu complexes il est
très facile d’écrire du code très peu performant avec nos commandes. J’ai
montré l’un de ces exemples plus tôt dans l’article. Dans ces cas il est
judicieux de revoir son code ou, si besoin, d’utiliser un nouvel outil.
Un très bon exemple de cela est documenté dans notre article sur sed et ses
performances. En somme, pour filtrer une fois sur une regex il est très
probable que grep soit le plus rapide mais s’il s’agit de l’enchaîner avec
deux ou trois autres opérations d’une manière un peu sophistiquée faites
attention.
- Libres
Ces logiciels sont tous libres. Je n’ai pas envie de prendre ici le temps d’expliquer pourquoi l’utilisation de logiciel et de formats libres devrait être la toute première étape lorsque l’on souhaite produire de la recherche reproductible.
Pour aller plus loin
Pour voir des exemples plus avancés vous pouvez jeter un coup d’oeil à cette tentative de reproduction d’un article analysant des données à propos d’un tournoi de starcraft.
Bêtisier
Quelques graphs un peu jolis mais inutilisables :
oups - 40Ko
oups le retour - 20Ko
-
Tabulation Seperated Data, du CSV mais séparé par une tabulation plutôt qu’un autre délimiteur. ↩
-
Pour translate ↩
-
Ici \t veut dire une tabulation. La commande exprime donc “traduit toutes les virgules en tabulation du fichier population.csv et met le résultat dans population.tsv” ↩
-
Pour insérer une tabulation plutôt que de provoquer l’autocomplétion vous pouvez faire ctrl+v puis appuyer sur la touche tabulation ↩
-
pour être honnête je n’ai pas fait beaucoup de graphs dans ma vie mais du peu que j’ai vu, aucun outil de graph n’est vraiment facile d’utilisation. ↩
-
A part les outils tsv-utils et gnuplot, tous les outils que nous voyons ici sont installés par défaut sur (presque) toutes les distributions linux. Gnuplot ne pèse “que” 3,5Mo ↩
-
J’aurais tendance à penser que dans ce cas particulier, si c’était le seul traitement que nous avions à faire alors ça ne vaudrait pas forcément le coup ↩
-
et c’est en réalité toujours le cas ↩
-
petite mise en pratique improptu hop ↩